数据可视化(EDA)
1、对数据集进行EDA是什么?
1、对数据集进行EDA是什么?
- EDA是一种用于分析数据集以概括其主要特征的方法,通常使用统计图形和其它数据可视化方法,是理解和准备用于任何数据集分析或机器学习项目的关键步骤。
2、要达成的主要目标是什么?
- 了解数据集的整体结构和分布。
- 识别数据中的异常值或错误。
- 发现数据中的模式和趋势。
- 为进一步的分析做好准备。
3、EDA常用的方法有哪些?
- 统计描述:使用统计量,例如均值、中位数、标准差来描述数据集的基本特征。
- 数据可视化:使用图标和图形来直观的展示数据,如直方图、箱线图、散点图等。
- 相关性分析:考察不同变量之间的关系。
- 降维:将高维数据降维到低维空间以便于分析。
4、数据分析技巧
- 检查数据的基本特征:
- 检查数据表的前几行:head()
- 检查数据类型和缺失值:df.info()
- 查看统计信息:df.describe()
- 处理缺失值、异常值和重复项:
- 对缺失值使用插补等技术
- 适当的识别和处理异常值
- 如果有必要直接删除某些样本
- 数据可视化:
- 创建可视化图表以深入洞察数据
- 对数据特征使用直方图和箱线图
- 对类别特征使用条形图
- 通过相关矩阵和散点图来理解变量间的关系
- 特征分析:
- 探究分类和异常检测中特征于目标变量之间的关系
- 通过可视化方法来展示特征在不同类型和类别键的差异和变化
- 利用箱线图、提琴图或蜂窝状堆积图来比较特征分布
5、数据EDA的例子
- 以UBC数据集为例子
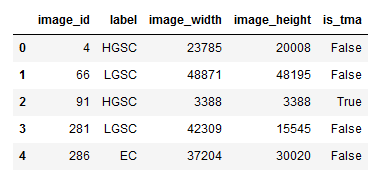
- 原始数据集长这样
- 一个csv文件包含图像名字和标签以及该图像大小等信息
- 一个文件夹里有对应名字的图像
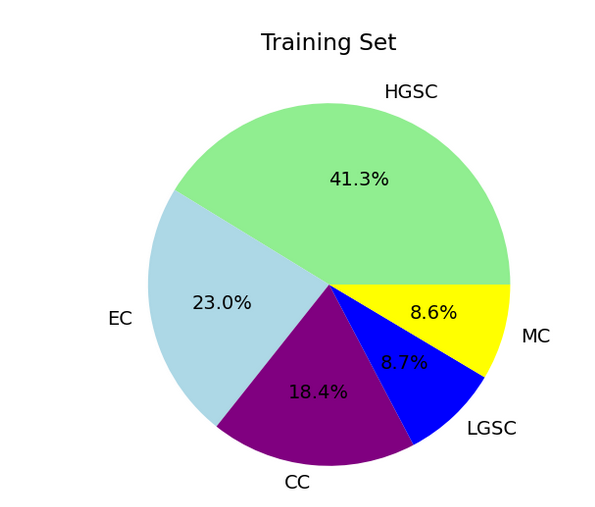
5.1 显示每个类别有多少训练数据
1 | train_df['label'].value_counts(): |
饼形图显示:
1 | # 查看所有标签类在数据集中所占的比例 |
5.2查看缺失值
1 | train = pd.read_csv('train.csv') |
5.3查看每个特征中有哪些不同的值
- 比如性别有男女,年龄有很多种等
1
2
3
4
5
6
7# 先提取保存不同的特征及其所不同的类别
feature_uniques = []
for cat in features:
feature_uniques.append(len(train[cat].unique()))
uniq_values_in_categories = pd.DataFrame.from_items([('name', features), ('unique_values', uniques)])
1 | fig, (ax1, ax2) = plt.subplots(1,2) |
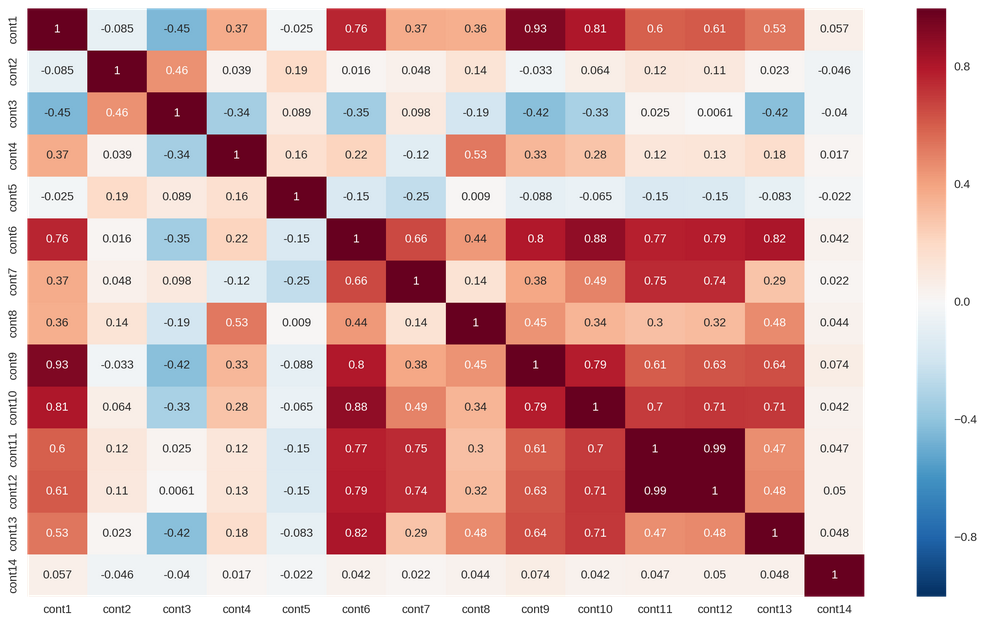
特征之间的相关性
1 | plt.subplots(figsize=(16,9)) |